摘要: 自然环境下植物根系具有丰富的微生物资源,这为植物生长和抗逆提供了重要的支持。本文提供了一套田间甘蔗根系样品采集和对甘蔗根系细菌进行高通量分离培养与鉴定的方法。研究中对田间甘蔗根系进行采集并对甘蔗根系的细菌进行高通量培养,采用双侧标签PCR扩增法高通量扩增并富集培养细菌16S rRNA基因的DNA,结合Illumina Novaseq 6000 PE250测序技术和生物信息学分析方法解析甘蔗根系细菌种群的组成。我们从甘蔗根系组织以及附着在根系表面的根际土中均分到9,000个左右的菌株,分析结果显示,来源于根系组织的细菌菌株归属于261个独特的扩增子序列变异体(ASV),相比之下,来源于根际土壤中的细菌菌株归属于457个不同的ASV,多样化程度更高;这些ASV主要属于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)。本文采用的方法为甘蔗根系微生物组资源库的建立提供了途径,为进一步研究根系细菌与甘蔗的互作关系以及防控甘蔗病害提供微生物资源。
关键词: 甘蔗, 根系微生物组, 高通量分离培养, 高通量鉴定
材料与试剂
- 甘蔗根系样品(采自华南农业大学增城教学科研基地)
- 丁腈手套
- 50 mL离心管
- 标签纸
- 滤纸
- 称量纸
- 无菌石英砂
- PCR引物(生工)
- 无水乙醇(天津市大茂化学试剂厂,产品编号:2335)
- PBS缓冲液(生工,产品编号:E607008-0500)
- 无核酸酶水(Qiagen,产品编号:129115)
- 13 × 13 cm方形培养皿(Biosharp,产品编号:BS-130-SD)
- 微量离心管(1.5和2 mL;Biosharp,产品编号:BS-15-M和BS-20-M)
- 胰蛋白胨大豆肉汤培养基(TSB,环凯微生物,产品编号:024051)
- 移液器枪头(10、200和1000 μL;Biosharp,产品编号:BS-10-T,BS-200-T,BS-1250-T)
- 96孔培养板(Biofil,产品编号:TCP010096)
- 96孔PCR板(Biosharp,产品编号:BS-PC96-C)
- 96孔PCR板封板膜(Labselect,产品编号:SF-SP-100A)
- 甘油(Macklin,产品编号:56-81-5)
- Escherichia coli DH5α(全式金,产品编号:CD601-02)
- PCR反应体系试剂(Takara,产品编号:R007Z)
- DL2000 Plus DNA Marker(诺唯赞,产品编号:MD101-01)
- Goldview核酸染料(Biosharp,产品编号:BS357A)
- 胶回收纯化试剂盒(Omega Bio-tek,产品编号:D2500)
- 氢氧化钠(Macklin,产品编号:S835850)
- 乙二胺四乙酸二钠(生工,产品编号:6381-92-6)
- 三羟甲基氨基甲烷(Genview,产品编号:BT504)
- 磁珠(Agencourt AMPure XP beads,Beckman,产品编号:A63882)
- TE 缓冲液(北京鼎国昌盛生物技术有限责任公司,产品编号:NEP029)
仪器设备
- 锄头
- 剪刀
- 记号笔
- 无菌研钵
- 研磨棒
- 无菌金属勺
- 冰盒
- 镊子
- 酒精灯
- 手术刀片
- 300 μL量程的8通道移液器(Eppendorf,产品编号:3125000052)
- 单道移液器(10、20、100、200和1,000 μL;Eppendorf,产品编号:3120000020、3120000038、3120000046、3120000054和3120000062)
- 生物安全柜(Esco,产品编号:AC2-6S1)
- 分析天平(赛多利斯,产品编号:2011F145-11)
- PCR仪(MiniAmp Plus Thermal Cycler;Thermo Fisher,产品编号:A37835)
- 96孔PCR板离心机(天根生化科技(北京)有限公司,产品编号:OSE-MP25)
- 电泳仪(北京六一生物科技有限公司,产品编号:DYY-6C型)
- 凝胶成像仪(Bio-Rad,产品编号:ChemiDoc XRS+ System)
- -20 ℃和-80 ℃超低温冰箱(海尔生物医疗,产品编号:DW-40L508J;DW-86L486)
- 高压蒸汽灭菌锅(Zealway,产品编号:GR60DA)
- pH计(上海仪电科技,产品编号:600313N00)
- 磁力架(Yeasen,产品编号:80461ES03)
- 微型离心机(Eppendorf,产品编号:5420000296)
- Nano-300微量分光光度计(杭州奥盛仪器有限公司,产品编号:AS-11020-00)
实验步骤
一、采样
- 用铲子去除地表植被和其他杂质,使用锄头轻轻刨开土壤,挖出甘蔗根部,轻轻抖落粘附在根上的土壤颗粒,保留附着在根系表面薄薄的一层甘蔗根际土。用消毒的剪刀剪取甘蔗根系组织,放入无菌的50 mL离心管中(图1),随即放入冰盒,完成样品采集。
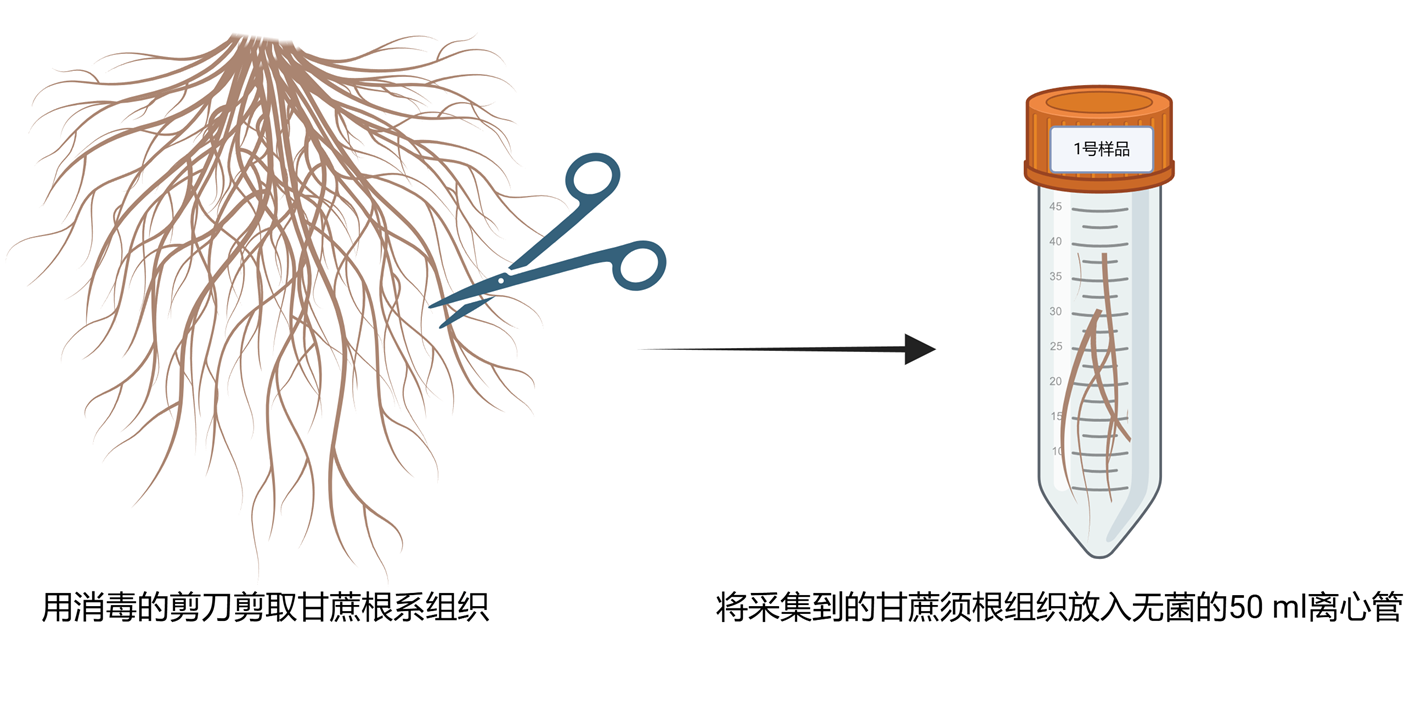
图1. 甘蔗根系组织的采样方法. 注:图片通过Biorender(https://BioRender.com)生成
二、样品处理
- 使用分析天平称取0.5 g根系组织样品,用无菌剪刀将样品剪成小段后转入新的50 mL无菌离心管中,加入2 mL无菌水浸没根系组织样品。将装有根系组织样品的50 mL离心管放入超声清洗仪中超声处理15 min,温度设置为25 ℃,期间轻轻晃匀管内样品2–3次,洗下根际土(图2)。
- 使用无菌镊子将超声处理后的根系组织样品夹出,收集根际土浸出液。将根际土浸出液在2,000 rpm(4 ℃)条件下离心30 min,吸取上清存于2 mL离心管中,获得根际土原液,置于冰盒中备用。
- 将处理后的根系组织样品转移至加有无菌PBS缓冲液的方皿中清洗干净(可重复2–3次),接着将样品转移至无菌滤纸上吸干表面水分。
- 准备无菌石英砂和无菌研钵,将样品和无菌石英砂放进无菌研钵中,加入PBS缓冲液,用无菌研磨棒研磨为匀浆状(此过程可分次加入总共4–8 mL PBS缓冲液)。研磨完毕后,将匀浆液分装入2 mL管中,在2,000 rpm(4 ℃)条件下离心30 min,并吸取离心后上清液置于新的2 mL离心管中,获得根系组织样品研磨原液,置于冰上备用。

图2. 甘蔗根系组织处理流程图. A. 根系组织样品的采集;B. 加入无菌水将根系组织样品浸没;C. 根际土原液;D. 获得根系组织样品;E. 研磨后的甘蔗根系组织样品;F. 根系组织样品研磨原液
三、稀释液的配置以及培养
- 将得到的根系组织研磨原液用TSB培养基稀释到原来浓度的十分之一,得到10-1的稀释液,即300 μL原液加入2.7 mL的TSB培养基;对10-1的稀释液进行进一步稀释,获得10-3的稀释液,即2 ml的10-1的稀释液加入到198 mL TSB培养基。
- 将根际土原液按照步骤1中稀释根系组织研磨原液的方法进行稀释,得到10-4的根际土稀释液。
- 摇匀稀释液,将稀释液倒入13 × 13 cm的无菌方皿中,用8通道移液器向96孔培养板的每个孔中转移120 µL稀释液,每瓶稀释液转移16个96孔培养板,每个96孔培养板的最后一列加入TSB培养基作为对照,并与稀释液之间空一列以防止稀释液飞溅污染对照组。转移完毕后用封口膜缠绕96孔培养板的边缘封板,并在板盖上注明样品名称和编号(图3)。
- 将所有封好的96孔培养板转移至30 ℃的环境下静置培养1–2周,期间观察96孔培养板内培养基是否浑浊,若培养基仍处于澄清状态则继续培养并监测其浑浊程度,若培养基呈现肉眼可见的浑浊状态即可进行菌株的保存及鉴定。根系组织10-3的稀释液、根际土10-4的稀释液静置培养一周后96孔培养板内每个孔都会浑浊。
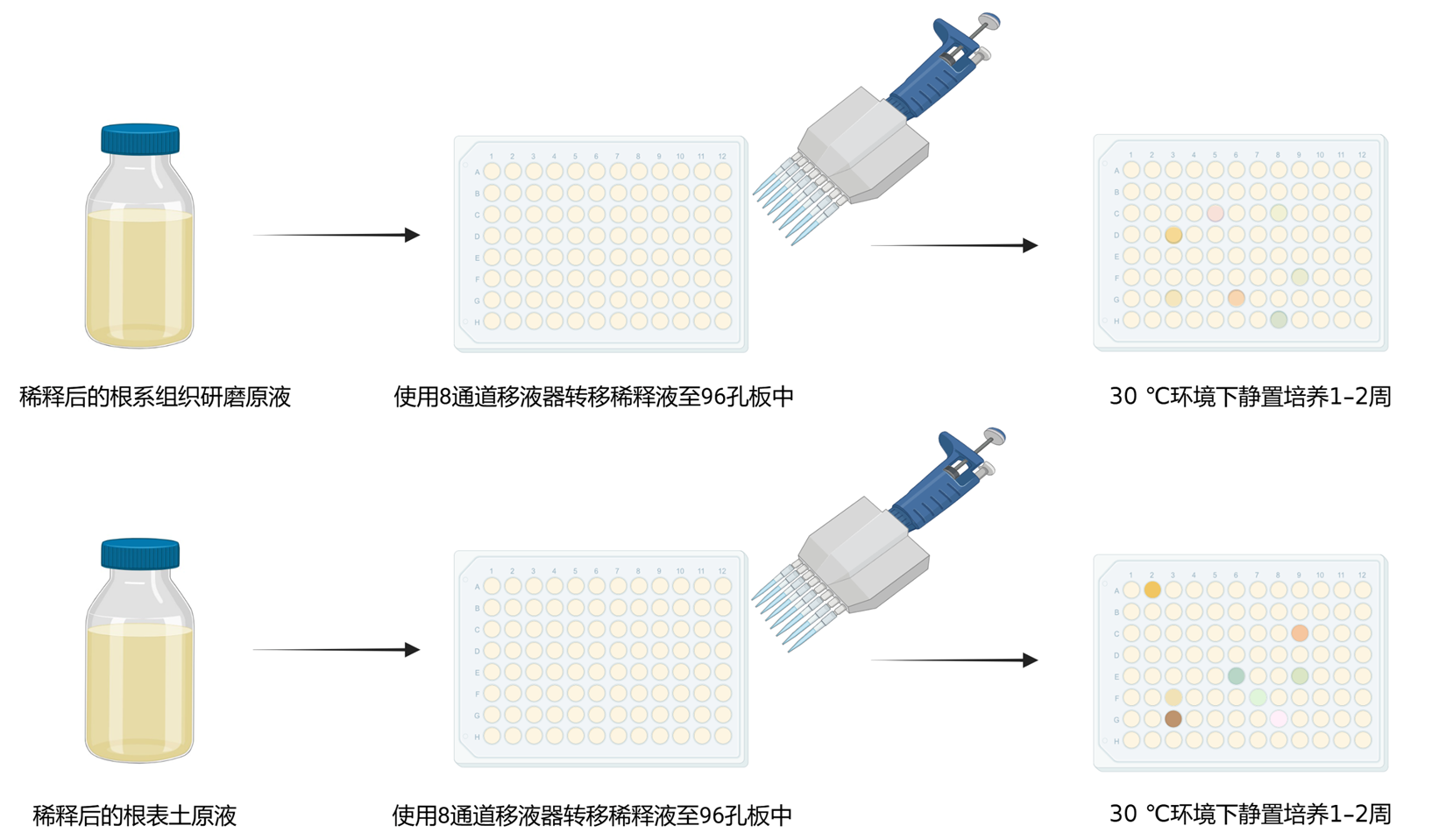
图3. 甘蔗根系样品稀释液的培养. 图片通过Biorender(https://BioRender.com)生成
四、鉴定
- 样品制备与保存:使用8通道移液器从96孔培养板内吸取10 μL培养获得的菌液,并将菌液转移到96孔PCR板中,PCR板中的A12和B12孔的样品为无核酸酶水和E.coli DH5α的基因组DNA(每个板上的阴性和阳性对照);使用封板膜对96孔PCR板进行封板,并将封闭好的96孔PCR板存储在-20 ℃的冰箱中用于后期的细菌鉴定。向96孔培养板中剩余的菌液中加入等体积的80%灭菌甘油,存储在-80 ℃冰箱中。
- 样品预处理:在细菌鉴定时,取出冻存在-20 ℃冰箱中的96孔PCR板,用96孔PCR板离心机短暂离心;在生物安全柜中,向每个孔中加入16.6 μL碱性裂解液与孔中的10 μL样品混合均匀,并使用封板膜进行封板。对封闭后的96孔PCR板进行离心,并将样品置于PCR仪进行高温裂解,程序设置为95 ℃,30 min;裂解结束后将96孔PCR板冷却降温,随后向每个孔中加入16.6 μL中和缓冲液,混匀并离心,完成预处理,每孔中完成预处理的产物将作为第一轮PCR检测使用的模板,完成预处理的产物可存储于-20 ℃冰箱。
- 样品鉴定:采用双侧标签两步法扩增样品16S rRNA基因。使用两对引物用以区分同一文库中不同样本的标签(Barcode)序列及Illumina测序所需序列,引物结构如图4所示。
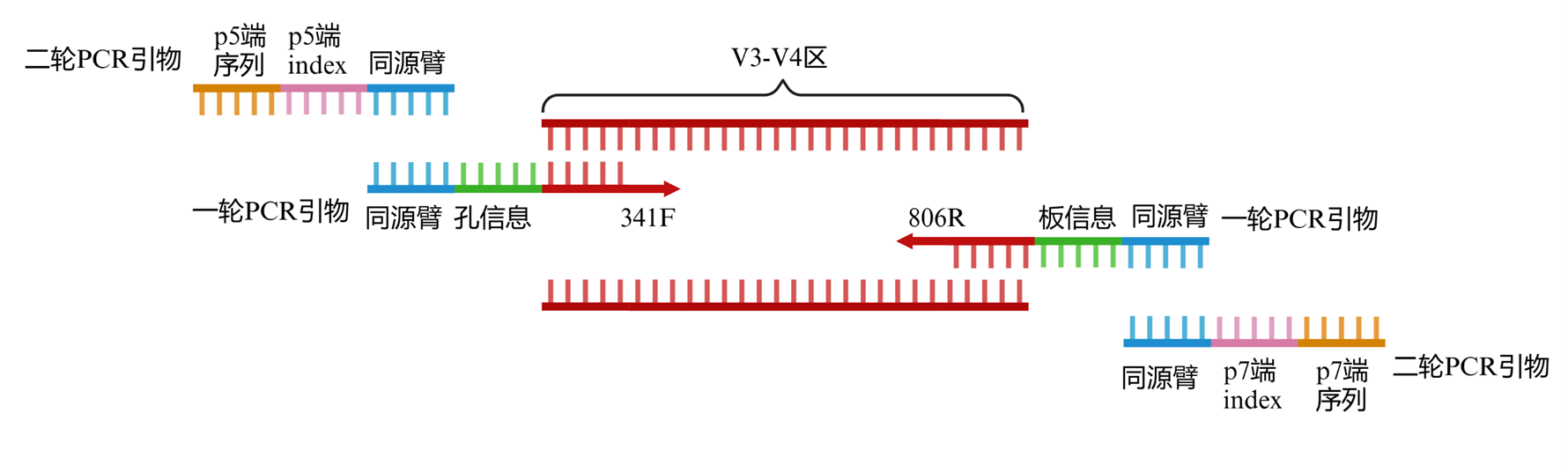
图4. 16S rRNA基因两步扩增所用引物的结构. 图片通过Biorender(https://BioRender.com)生成
- 第一轮PCR检测使用的引物见附表1中的一轮PCR引物。一轮PCR引物是通过在16S rRNA基因的通用扩增引物341F和806R的5’端分别加入不同的Barcode序列(用以区分文库中的不同样本)来构建,F端引物共96条,用于标记96孔培养板中的每个孔。R端引物共48条,用于标记每个文库中不同的96孔培养板,每个文库可承载48 × 96个不同的样本。第一轮PCR检测的扩增体系和程序分别见表1和表2所示。
表1. 一轮PCR体系
| 成分 | 体积(μL) |
| 10× buffer | 3 |
| dNTPs(2.5 mM each) | 2.4 |
| 二轮PCR引物-F | 0.6 |
| 二轮PCR引物-R | 0.6 |
| HS taq(5U/μL) | 0.15 |
| 模板 | 3 |
| 无核酸酶水 | 20.25 |
| 总计 | 30 |
表2. 一轮PCR程序
| 时期 | 温度 | 时间 | 循环数 |
| 预变性 | 94 ℃ | 2 min | 1 |
| 变性 | 94 ℃ | 30 s | 30 |
| 退火 | 55 ℃ | 30 s |
| 延伸 | 72 ℃ | 1 min |
| 后延伸 | 72 ℃ | 5 min | 1 |
- 取3 μL第一轮PCR的阴性和阳性对照产物,分别混合适量的loading buffer进行电泳检测。若检测结果为阳性对照产物的条带约400 bp,阴性对照产物无条带,则表明第一轮PCR反应合格。
- 第二轮PCR使用二轮PCR引物(附表2),该引物为第二轮PCR产物加上文库索引(index)序列及测序接头。将第一轮的PCR产物稀释为原来浓度的四十分之一作为第二轮PCR模板,A12和B12孔的样品同样作为阴性和阳性对照。扩增体系如表3所示,扩增程序如表4所示。
表3. 第二轮PCR体系
| 成分 | 体积(μL) |
| 10× buffer | 3 |
| dNTPs(2.5 mM each) | 2.4 |
| 二轮PCR引物-F | 0.6 |
| 二轮PCR引物-R | 0.6 |
| HS taq(5U/μL) | 0.15 |
| 模板 | 3 |
| 无核酸酶水 | 20.25 |
| 总计 | 30 |
表4. 第二轮PCR程序
| 时期 | 温度 | 时间 | 循环数 |
| 预变性 | 94 ℃ | 2 min | 1 |
| 变性 | 94 ℃ | 30 s | 25 |
| 退火 | 55 ℃ | 30 s |
| 延伸 | 72 ℃ | 1 min |
| 后延伸 | 72 ℃ | 5 min | 1 |
- 吸取5 μL第二轮PCR的阴性和阳性对照产物,分别混合适量的loading buffer进行电泳检测。阳性对照产生约500 bp的条带,阴性对照无条带,表明第二轮PCR反应合格。
- 将每个PCR板中的产物转移至1个5 mL无菌离心管中混匀,取40 μL混合产物与适量loading buffer混合,使用1.2%琼脂糖凝胶,在120 V/cm的条件下进行电泳40 min。
- 干净的手术刀片切取含有500 bp大小DNA片段的琼脂糖凝胶块(尽量减小胶块的体积),并将琼脂糖凝胶块转移到已称好重量的1.5 mL离心管内,同时记录凝胶的重量。
- 使用胶回收纯化试剂盒对琼脂糖凝胶块进行回收纯化,回收的DNA溶液储存在新的1.5 mL离心管中,使用Nano-300微量分光光度计测定浓度后保存在-20 ℃冰箱。
- 将每个PCR板获得的DNA产物各取70 ng转移到一个1.5 mL离心管中,混合均匀。
- 使用磁珠法对混合后的DNA产物再次进行纯化和浓缩,将磁珠提前从4 °C冰箱中取出,室温孵育30 min,按照磁珠加入量:样品体积 = 0.9:1的比例,向混合后的DNA样品中加入磁珠,室温静置10 min。
- 将1.5 mL离心管置于磁力架吸附5 min,磁珠被吸附至离心管的侧壁,用移液器去除上清(去除上清时应尽量避免吸到磁珠,减少DNA的损失)。
- 加入200 µL现配的80%乙醇,将离心管侧壁吸附的磁珠完全浸没,孵育30 s后,去除乙醇。重复此步骤1次。
- 将磁珠置于室温干燥约4 min,待磁珠表面的液体层消失后,完成对磁珠上残留乙醇的去除。
- 撤去磁力架,向每个离心管中加入100 µL的1× TE缓冲液,混合均匀后室温静置5 min,洗脱磁珠上的DNA。
- 将离心管重新置于磁力架吸附5 min,吸取96 µL上清液至新的1.5 ml无菌离心管,完成第一次纯化浓缩。
- 为了进一步纯化和浓缩PCR产物,在第一次纯化浓缩的样品中添加86.4 µL磁珠,混匀后室温静置10 min,随后置于磁力架吸附5 min,最后用移液器去除上清液。加入200 µL现配的80%乙醇,孵育30 s后用移液器去除乙醇,此步重复1次。室温干燥约4 min去除残留乙醇。
- 撤去磁力架,加入50 µL的1× TE缓冲液洗脱DNA,用移液器上下吹打30次,室温静置5 min。
- 置于磁力架吸附5 min,吸取48 µL上清液于新的1.5 ml无菌离心管。
- 回收产物使用Nano-300微量分光光度计定量。
- 取1,500 ng的纯化PCR产物进行Illumina测序,测序服务由北京诺禾致源科技股份有限公司提供。剩余文库样品置于-20 ℃保存。
- 生物信息学分析鉴定分离到的细菌
本研究基于Zhang等[2]开发的高通量细菌鉴定流程,进行了个性化定制以完成本课题细菌培养物的鉴定,实现甘蔗根系细菌培养物的双端标签化扩增子文库的分析和可视化全流程。
在本研究中,数据分析流程综合运用了多种编程语言(包括R、Shell、Python和Perl)编写的脚本,并通过RStudio集成开发环境执行。为确保分析环境的一致性和其依赖包的有效管理,我们采用Conda软件管理器Miniconda3来创建和维护虚拟环境。此外,我们的分析还依赖于微生物组学分析常用的软件工具中的相关功能,以支持深入的数据解析和生物信息学分析。例如,QIIME被用来根据序列标签拆分样本;VSEARCH则负责引物切除、去冗余及基于RDP(Ribosomal Database Project)数据库的物种注释工作。
另外,我们安装了一系列关键R包用于数据处理后的可视化,包括如柱状图、箱线图以及稀释曲线的绘制,热图、物种分类树的生成等。例如,统计每个板和孔中的测序深度,并绘制其柱状图观察每板的测序量是否均匀。详细代码如下:
mkdir -p result/split
for l in `cat result/library.txt`; do
tail -n+16 temp/${l}/split_library_log.txt| head -n-4 \
> result/split/${l}.txt
stat_split_bar.R \
-i result/metadata.txt -d result/split/${l}.txt \
-o result/split/
done
为了排除实验过程中的污染以及由于测序错误而造成的假阳性的结果,对 96孔培养板中的阴、阳性对照的数据量进行计算,确定过滤假阳性的阈值。每个板A12是无核酸水的负对照,B12是E.coli DH5α的DNA正对照。
negative_threshold.R \
--input temp/ASV_table.txt --metadata result/metadata.txt \
--threshold 1 --negative A12 --positive B12 \
--output result/fdr.txt
鉴定每个ASV序列对应的候选菌株,评估ASV多样性与工作量之间的饱和度。
time identify_isolate.R \
--input result/ASV_table.txt \
--genus result/genus.count \
--taxonomy result/taxonomy_8.txt \
--output result/isolate
绘制96孔培养板的纯度热图。
mkdir -p result/purity
for l in `cat result/library.txt`; do
# Prepare input file
awk 'NR==FNR{a[$1]=$3}NR>FNR{print $1"\t"a[$1]}' \
result/isolate_well.txt seq/${l}.txt | tail -n+2 | sed 's/\t$/\t0/' \
> result/purity/${l}.txt
# Format list into plate format
format_list2plate.pl -i result/purity/${l}.txt \
-o result/purity/${l}/
# Batch plot top 1 plate, deleted '|head -n1' to plot all
list=`ls result/purity/${l}/|cut -f 1 -d '.'|cut -f 2 -d 'P'|sort|uniq|head -n5`
for plate in $(echo $list);do
plot_pheatmap.sh -i result/purity/${l}/${l}P${plate}.plate \
-o result/purity/${l}/${l}P${plate}.png
done; done
使用GraPhlAn绘制培养菌物种进化分支图。
graphlan_prepare.R --input result/ASV_table.txt \
--taxonomy result/taxonomy_8.txt \
--abundance 0 --number 250 \
--output result/graphlan/
graphlan_plot.sh -i $db -o result/graphlan
结果与分析
在高通量分析培养甘蔗根系细菌的过程中,我们对甘蔗根系组织、根际土分别设置了三组重复。培养2周后,根系组织以及根际土两组样品的96孔培养板中均表现出有90%以上的孔出现浑浊的情况。对每个板的孔绘制纯度热图,未有细菌生长的孔(即未出现浑浊的孔)纯度为“0”,其余孔的纯度反映孔中细菌的多样性,数值越接近100表明孔中细菌的种类越单一(图5)。
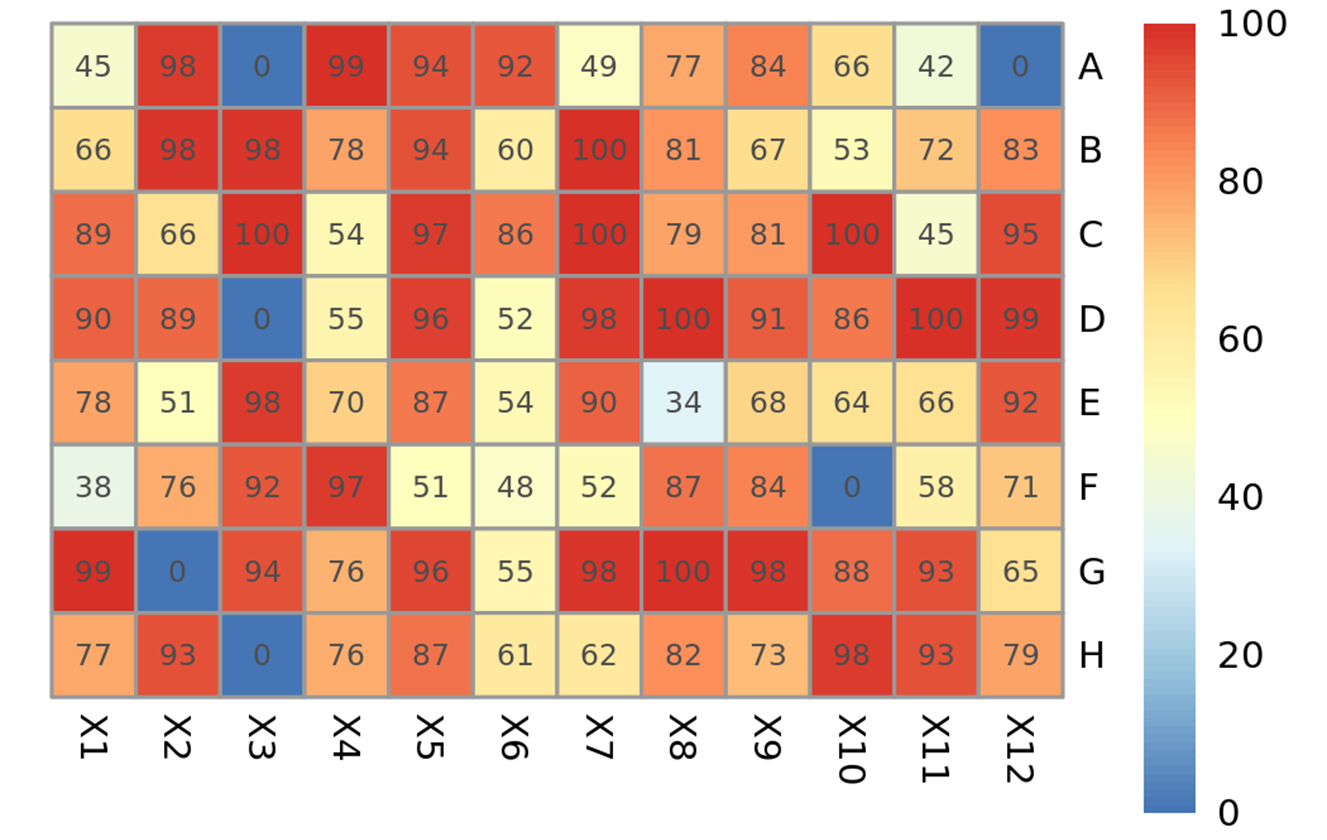
图5. 96孔培养板中每个孔的纯度热图(以P06板为例). 基于ASV表和物种注释得到孔信息列表,根据每个孔中的前三个ASV绘制纯度热图。
对所有96孔培养板测序数据的读段数量进行统计,结果表明,每一个96孔培养板的测序读段数量保持高度的一致,约为21696条序列,没有出现明显的测序深度过高或过低的现象(图6)。这种均匀的测序深度分布不仅保证了每个样本区域都能被充分且均衡地覆盖,而且能够高效和无偏差的鉴定培养的细菌。
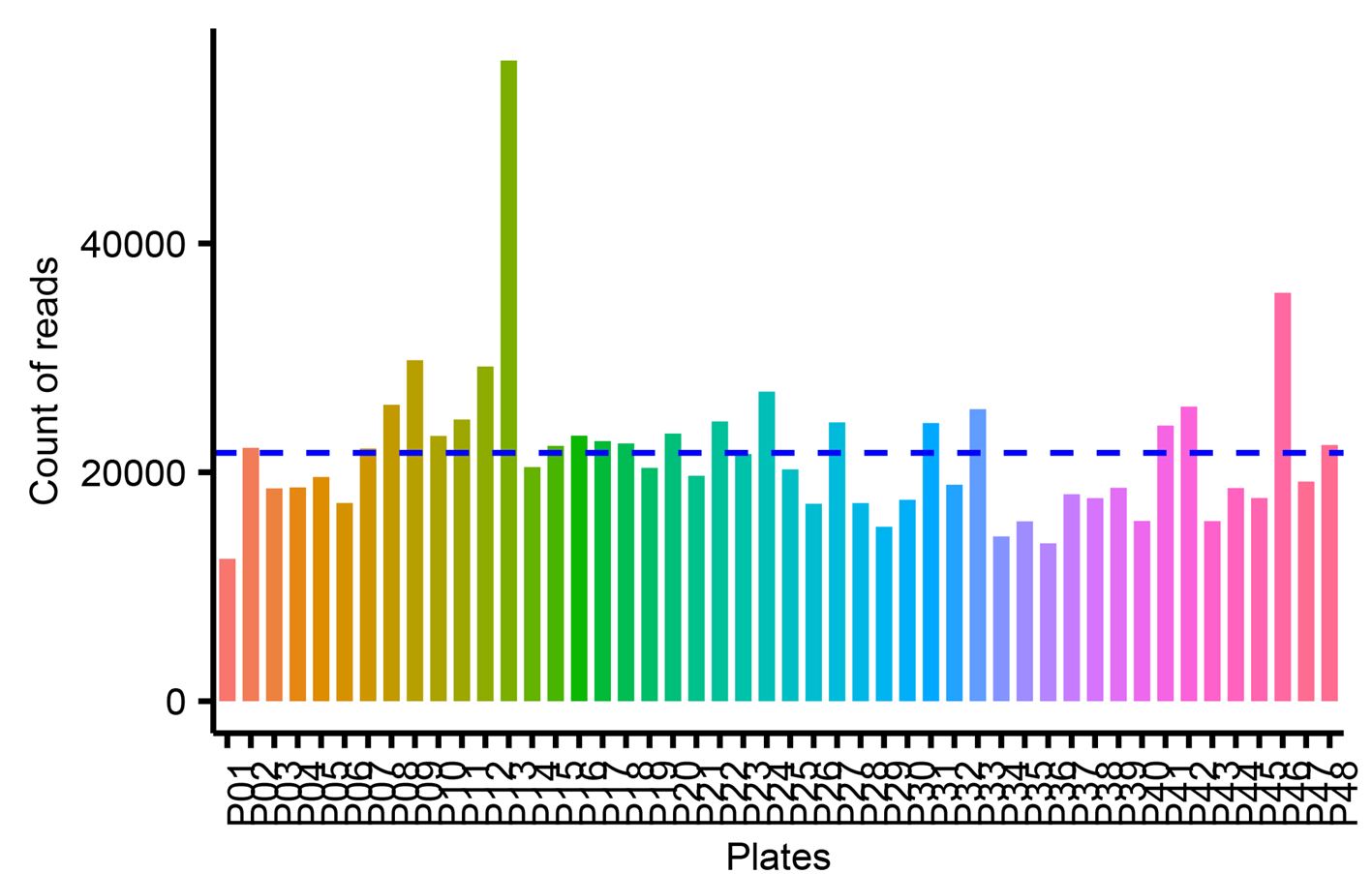
图6. 每个96孔培养板读长数量的柱状图
在PCR过程中由于测序技术敏感以及所用耗材、试剂中的污染也会被扩增而放大信号。为了确保实验准确性并减少假阳性结果的影响,我们采用了阴性对照过滤的方法,对每个细菌培养板上的阴性对照进行了检测。检测结果表明,阴性对照孔中的序列读取量几乎都低于18,证明了PCR鉴定体系的有效性和可靠性(图7)。
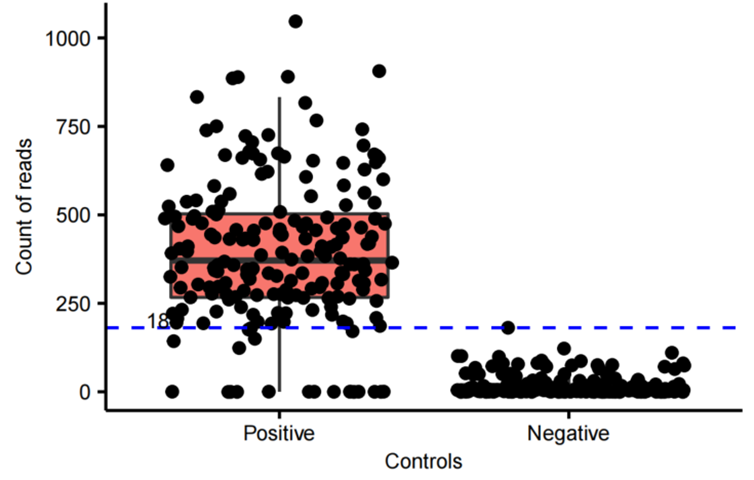
图7. 正对照(E.coli DH5α的基因组DNA)和负对照(无核酸酶水)的数据量分布箱线图
在对测得的细菌种类进行分析后我们注意到,随着培养板数量的增加,所分离出的细菌种类多样性也逐步上升,最终达到一个平稳状态,这表明分离出的细菌数量已接近饱和(图8)。这一结果表明,在当前设定的培养条件下,我们已经成功分离到了相当丰富的细菌种类,继续扩大分菌工作可能不会显著增加新菌种的发现概率。
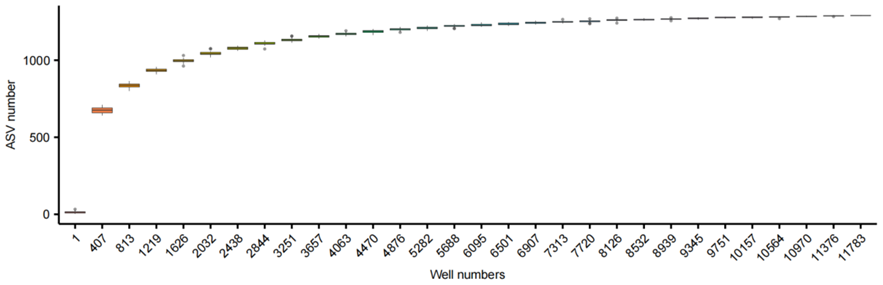
图8. 基于测序孔与ASV数量的稀释箱线图
最后我们从甘蔗根系组织以及甘蔗根际土样本中分别分离到了261和457个不同的ASV,这些ASV主要属于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)(图9)。
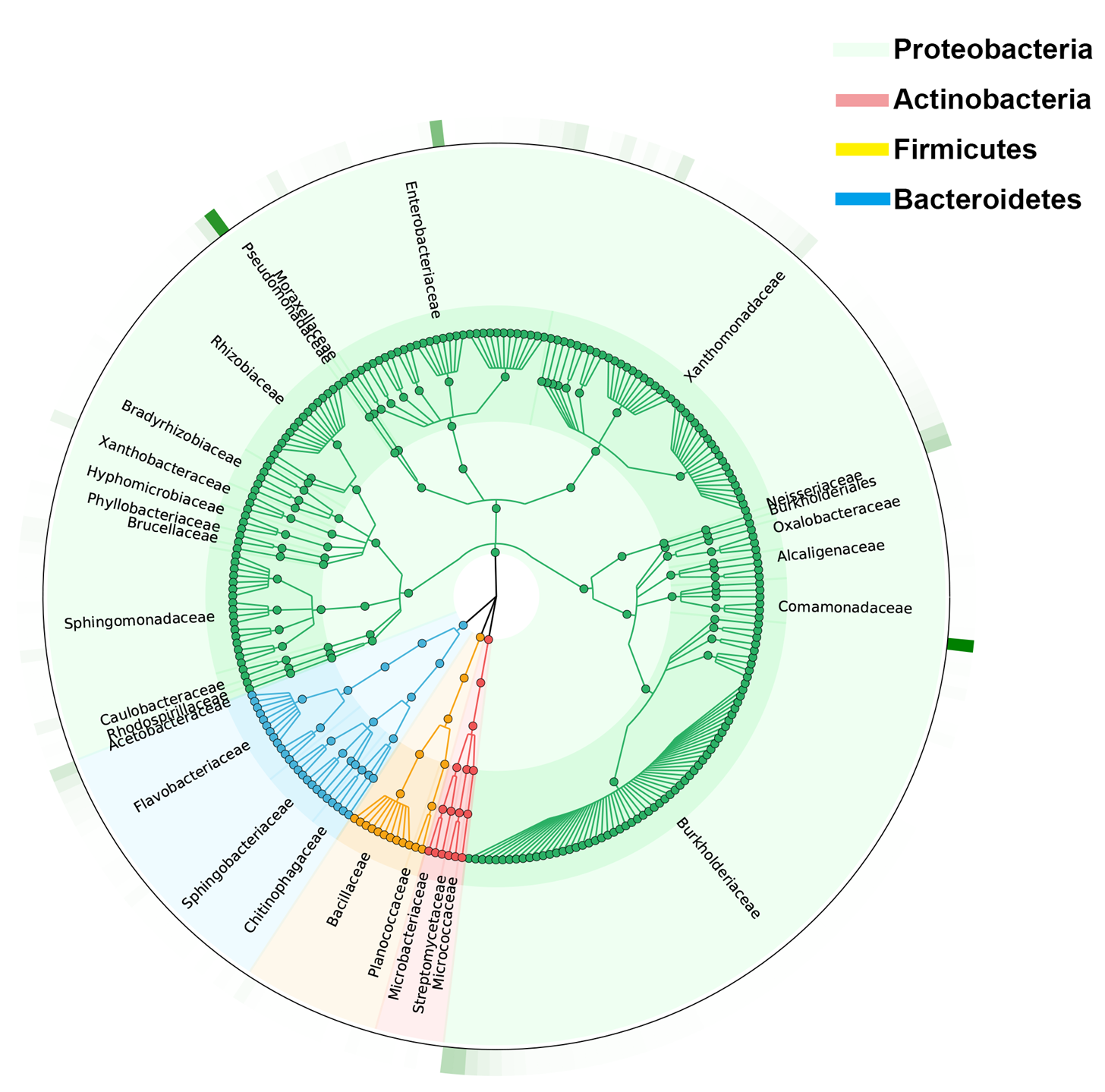
图9. 物种树展示培养菌的分类学组成(以根系组织培养菌为例)。不同颜色代表来源于不同门的细菌。
高通量分离培养鉴定根系微生物细菌方法的目标是从植物根系中高效分离可培养的微生物,结合高通量测序技术与生物信息学分析法高效鉴定分离获得的微生物,建立菌种资源库。该方法可以系统分离、培养和鉴定植物根系微生物,在菌株分离效率和种属覆盖度上,与传统组织样本分离方法(稀释涂布获得纯培养菌并逐一进行鉴定)相比具有显著差异和优势。Zhang等采用的高通量分离培养鉴定法,可在一天内完成组织样本的微生物分离工作,传统分离方法可能需要几周时间才能完成组织样本微生物的分离[2]。将细菌菌悬液稀释并在96孔培养板进行培养,可以避免快速生长细菌挤占慢速生长细菌的生长空间,进一步提高组织样本细菌的分离度。Zhang等通过高通量分离培养鉴定法在水稻根部分离到的278个ASV,占相关细菌分类单元的57.6%,相对丰度达78.4%,在种属覆盖度上有较好的表现[2]。
高通量分离培养鉴定根系微生物细菌方法已被证明可用于禾本科作物(水稻和甘蔗)根系微生物组的高通量分离与鉴定,但该方法是否可用于其它作物的根系细菌的高通量分离与鉴定,有待进一步研究。
失败经验
- 避免样本之间交叉污染。样品采集过程中不同样本易产生交叉污染,研究中需使用新的或消毒过的耗材(如手套、剪刀、镊子、铲子等)。
- 在28 ℃静置培养期间96孔培养板中部分孔内的培养基会蒸发。为防止孔内培养基蒸发损失,应在观察每个96孔培养板中菌液浑浊程度的同时,也观察孔内培养基体积,优先处理培养基浑浊且孔内培养基蒸发相对较多的96孔培养板。
- 当PCR板储藏于-20 ℃冰箱时,若封板膜未贴紧PCR板,封板膜容易开裂,无法牢固黏附在PCR板中。建议在PCR板上贴好封板膜后,使用刮板等工具使封板膜与PCR板紧密粘合。
- 在进行切胶回收实验离心时若离心力低于16,000× g(转速14,000 rpm)会导致产量降低。
溶液配方
- 碱性裂解液配方
25 mM NaOH、0.2 mM Na2-EDTA(pH = 12)。121 ℃灭菌20 min。冷却后储存在4 ℃环境下,保质期2个月。 - 中和缓冲液配方
40 mM Tris-HCl(pH = 7.5)。121 ℃灭菌20min。冷却后储存在4 ℃环境下,保质期2个月。 - 液体TSB培养基
将3 g TSB粉末溶解于1 L去离子水中,分装相应体积至试剂瓶中,121 ℃高压灭菌20 min。冷却后储存在4 ℃环境下,保质期1周。 - 75%(vol/vol)乙醇
250 mL去离子水与750 mL无水乙醇混合。 - 80%(vol/vol)甘油
200 mL去离子水与800 mL甘油混合。121 ℃高压灭菌20 min。 - 80%(vol/vol)乙醇
200 mL去离子水与800 mL无水乙醇混合。
致谢
本项目由国家自然科学基金区域创新发展联合基金(U23A20148)资助。
参考文献
- 尚宪超.(2022).防治烟草青枯病的根际核心微生物筛选鉴定与抗病机制.博士学位论文.中国农业科学院.
- Zhang, J., Liu, Y. X., Guo, X., Qin, Y., Garrido-Oter, R., Schulze-Lefert, P. and Bai, Y. (2021). High-throughput cultivation and identification of bacteria from the plant root microbiota. Nat Protoc. 16(2): 988–1012. https://doi.org/10.1038/s41596-020-00444-7
Please login or register for free to view full text
View full text
Download PDF
Q&A
Copyright: © 2025 The Authors; exclusive licensee Bio-protocol LLC.
引用格式:毕新萍, 杨翼航, 利晓彤, 胡志剑, 梁志彬, 邓懿祯. (2025). 甘蔗根系细菌高通量分离培养与鉴定技术. // 微生物组实验手册.
Bio-101: e1011033. DOI:
10.21769/BioProtoc.1011033.
 How to cite:
How to cite: Bi, X. P., Yang, Y. H., Li, X. T., Hu, Z. J., Liang, Z. B. and Deng, Y. Z. (2025). Techniques For High-throughput Isolation and Identification of Sugarcane Rhizospheric Bacteria. // Microbiome Protocols eBook.
Bio-101: e1011033. DOI:
10.21769/BioProtoc.1011033.
